관련 링크 : https://arxiv.org/abs/1505.04597
Segmentation : Unet(2015)
Abstract
Deep networks를 학습시키기 위해서는 수천장의 annotated training sample이 필요하다. 본 논문은 소량의 annotated sample에 data augmentation을 적용해 학습하는 네트워크를 제안한다. 이 네트워크는 context를 포착하는 contracting path와 localization을 위한 expanding path로 이루어지고, 적은 이미지로 end-to-end로 학습시켜 이전 방법들보다 좋은 성능을 보이면서 훨씬 빠르다.
1. Introduction
Deep network는 하나의 이미지당 하나의 class label을 가지는 classification task에서 좋은 성능을 보인다. 하지만, 대부분의 visual task는 localization task를 위해 각 픽셀당 class label을 예측해야 한다. Visual tasks 중 Biomedical image processing에서는 초기에 알기 원하는 픽셀 주변의 이미치 패치를 이용해서 class를 예측했다. 이 과정으로 localization이 가능해지고, 이미지 패치를 이용해 training image을 증식시키는 효과를 얻었다. 하지만 하나의 이미지를 segmentation하기 위해 수십 장의 patch를 이용한 계산(Sliding Window)해야 했고, context의 사용과 localization 정확도 사이의 trade-off가 생겼다. 다행히 이 후 연구에서는 이런 trade-off가 해소 될 수 있음이 밝혀졌다.
Sliding Window방식은 수십 장의 overlapping 패치를 이용해 모델이 한 부분을 여러번 보게 되므로 redundancy가 생긴다. 또 한 픽셀의 class 예측을 위해 넓은 패치를 사용하면 정확도를 감소시키는 maxpool로 이미지를 줄여야하고, 작은 패치를 사용하면 너무 작은 context를 보게 되는 trade-off 관계가 형성된다.
본 논문에서는 Fully convolutional network라 불리는 구조를 수정, 보완해 U자의 형태를 가진 U-net을 제안한다. 핵심 아이디어는 일반적인 contracting network에 pooling을 upsampling으로 대체한 expanding network를 추가하는 것이다. 따라서 최종 출력은 각 픽셀의 class를 가진 segmentation map이 되고, 따라서 fully connected layer는 필요가 없다.
이 외에도 여러 변경점 및 추가점들이 있고, 다음과 같다.
- 이미지의 최외각 픽셀들의 context를 보존하면서 어떤 크기의 이미지가 들어와도 적용 할 수 있게 overlap-tile strategy와 mirror padding을 사용한다.
- Network가 변형에 invariance하도록 elastic deformation을 사용한다.
- 동일한 물체들의 분리를 위해 weighted loss를 사용한다.
2. Network Architecture
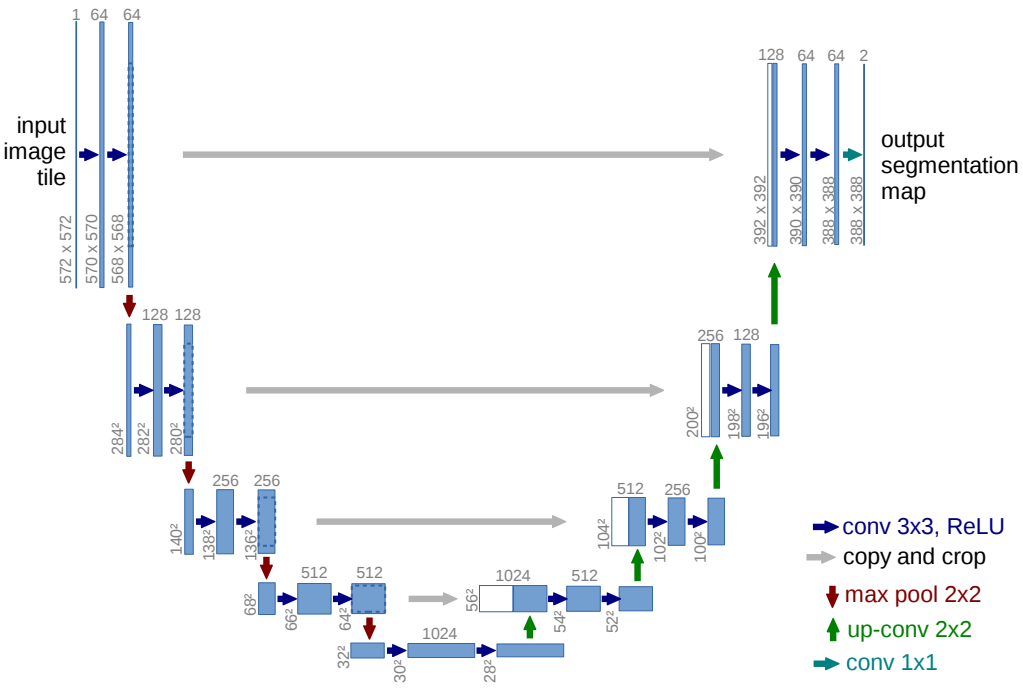
하얀색 상자는 contracting network에서 가져온 feature map이다.
-
Contracting path에서 unpadded convolution을 수행하므로 매번 feature map의 해상도가 줄어든다. 따라서, expanding path에서 이전 feature map을 concatenation할 때 cropping이 필요하다.
-
Segmentation map은 class의 개수만큼의 채널 수를 가져야 한다.
-
입력의 해상도에 관계없이 segmentation map을 만들기 위해 위에서 언급한 overlap-tile strategy와 mirror padding을 사용한다.
-
$2\times 2\;\mathrm{maxpool}$을 사용하기 때문에 입력의 해상도를 짝수로 만들어 줘야 한다.
3. Training
- SGD를 이용해 모델을 학습시킨다.
- 항상 출력이 입력보다 작으므로, GPU 사용 효율을 최대화하기 위해 입력 tile의 해상도와 batch size를 모두 키운다.
- 학습 속도를 키우기 위해 $0.99$의 높은 모멘텀값을 사용한다.
- He initialization을 이용한다.
Loss Function
Segmentation map은 class의 개수 $K$만큼의 채널을 가진다.
\[E=\sum_{\mathbf x \in \mathit\Omega}w(\mathbf x)\log(p_{\ell(\mathbf x)}(\mathbf x)) \tag 1\]-
$p_k(\mathbf x)=\exp(a_k(\mathbf x))/\left (\sum^K_{k`=1}\exp(a_{k’}(\mathbf x))\right)$
- $a_k(\mathbf x)$ : 각 feature map에서 픽셀의 위치 $\mathbf x \in \mathit \Omega$에서 $k$ 채널의 activation($\mathit \Omega \subset \mathbb Z^2$)
- $a_k(\mathbf x)$가 최대값을 가지는 $k$에서 $p_k(\mathbf x)\approx 1$
-
$\ell : \mathit\Omega \rightarrow\{1,\ldots,K\}$은 각 픽셀의 true class
-
위에서 언급한 weighted loss
\[w(\mathbf x)=w_c(\mathbf x)+w_0\cdot\exp\left (-\frac {(d_1(\mathbf x)+d_2(\mathbf x))^2}{2\sigma^2}\right) \tag2\]-
$w_c : \mathit\Omega \rightarrow \mathbb R$은 class frequency를 조절하기 위한 부분으로, 논문에 특별한 언급이 없다.
-
$d_1 : \mathit\Omega \rightarrow \mathbb R$, $d_2 : \mathit\Omega \rightarrow \mathbb R$은 각각 가장 가깝거나 두 번째로 가까운 cell의 경계까지의 거리
-
실험에서 $w_0=10$, $\sigma \approx 5$을 사용했다.
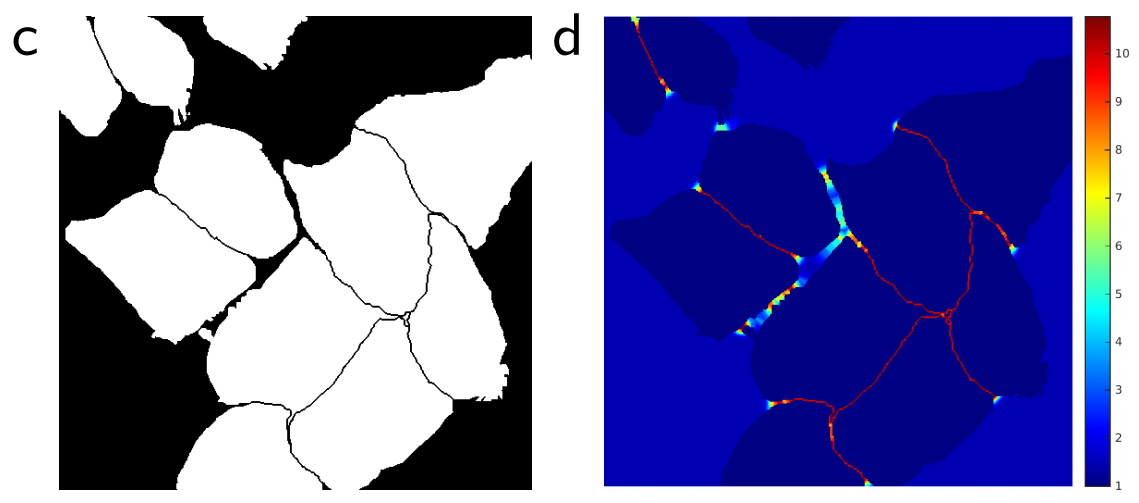
즉, 식 (2)의 두 번째 항은 픽셀이 경계에 있으면 작아지고 경계에서 멀어지면 커져서 경계 부근의 픽셀들이 큰 가중치를 얻도록 설계되었다. 아래 그림의 c는 생성된 segmentation mask, d는 각 픽셀의 loss map이다. 경계 부근의 가중치를 키워 loss가 크고, 모델이 성공적으로 경계면을 학습했다.
-
3.1 Data Augmentation
-
여러 변형들에 invariance하도록 data augmentation을 이용한다.
-
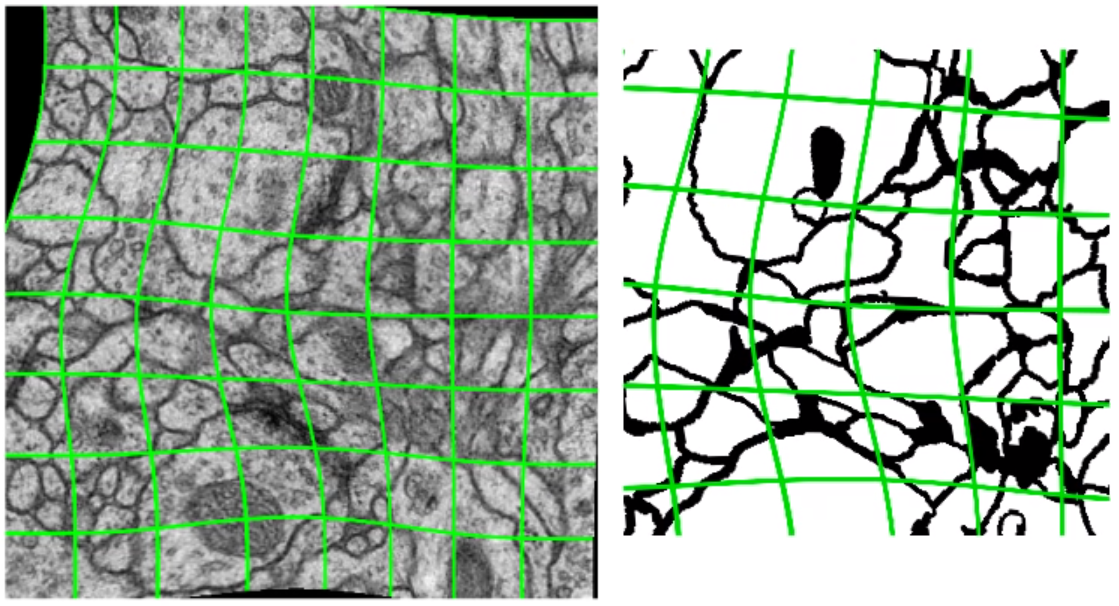
사용 가능한 training sample이 적으므로, random elastic deformation이 효과적이다.
의료데이터는 개개인마다 형태가 약간씩 다를 수 있다. 따라서 deformation에 invariance한 것은 중요한데, elastic deformation은 아주 효과적이다. 아래 사진은 elastic deformation의 예시 중 하나로, 시각화를 위해 shift, rotation, extraploation을 하지 않았다.
4. Experiments
EM segmentation challenge
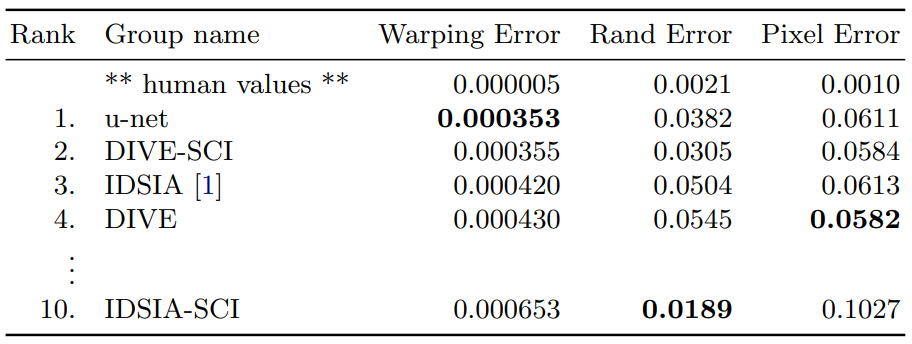
- DIVE-SCI 같은 경우는 사용된 데이터셋에 많은 post-processing을 적용했지만, u-net은 어떠한 pre- or post-processing도 적용하지 않았다.
- Sliding Window 방식을 이용한 IDSIA보다 성능이 좋다.
Warping Error란?
Segmentation metric중 하나로 topological Error를 의미한다. 아래 그림에서 A와 B의 pixel error는 같지만, B가 warping error가 더 작다.
ISBI cell tracking challenge
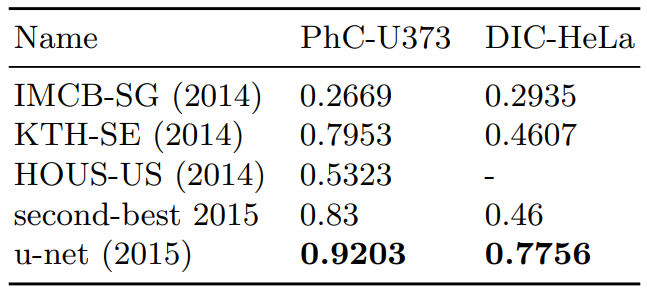
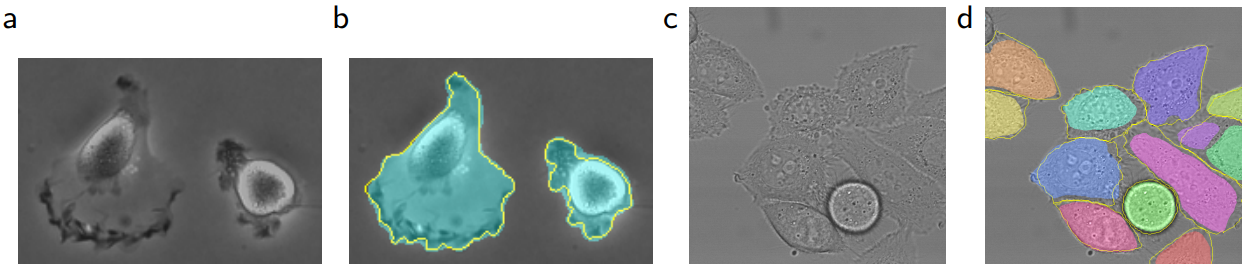
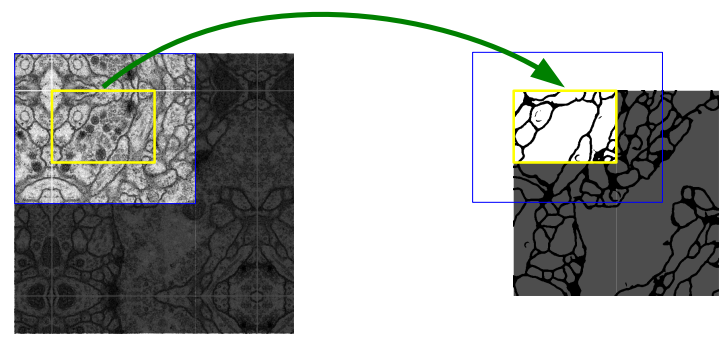
a, b는 PhC-U373, c, d는 DIC-HeLa 데이터 셋에서 Segmentation 결과이다. 노란선이 ground thruth이다.
- 두 번째로 성능이 좋은 방법보다 훨씬 좋다.
5. Conclusion
- 여러 biomedical segmentation에서 좋은 성능을 보인다.
- Elastic deformation을 이용해 아주 적은 training sample로 좋은 성능을 보인다.
- NVidia Titan GPU(6 GB)에서 10 시간정도의 짧은 시간으로 학습 할 수 있다.


Leave a comment